以细菌搜索结果为例:),目前只有这个结果还比较拿得出手
实验参数设置
| 参数 | 说明 |
|---|---|
| NoEnzyme U _ C | 非酶切方式,允许肽段在任意位置断裂,U和C为可能的断裂残基(非标准酶切位点)。 |
| MS/MS Tolerance | 20 ppm,质谱误差容忍度较小,说明仪器精度较高。 |
| 固定修饰 | Carbamidomethyl[C],半胱氨酸的烷基化修饰,防止二硫键形成。 |
| 可变修饰 | Oxidation[M],甲硫氨酸氧化,常见于氧化应激样本。 |
| 最大漏切数 | 0,非酶切模式下无漏切概念,设为0合理。 |
| 共洗脱(co elute) | True,启用共洗脱功能,提升低丰度肽段识别率。 |
| 开放搜索(open search) | True,允许未修饰肽段和修饰肽段同时搜索,适合未知修饰发现。 |
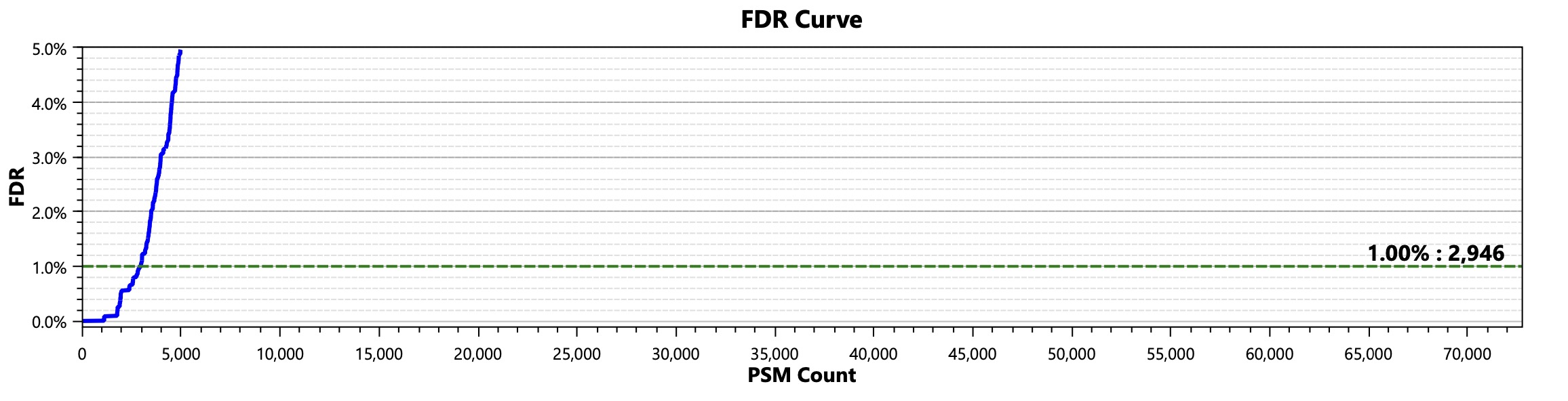
FDR 曲线与质量控制
- FDR(假发现率)曲线:
- 图中显示 FDR 控制在 1% 时,PSM(Peptide-Spectrum Match)数量约为 29,460。
- 说明数据质量较好,鉴定结果可信度高。
质量偏差(Mass Deviation)
- 平均偏差:约 ±0.0044 Da,说明质谱质量精度较高,符合 20 ppm 的设定。
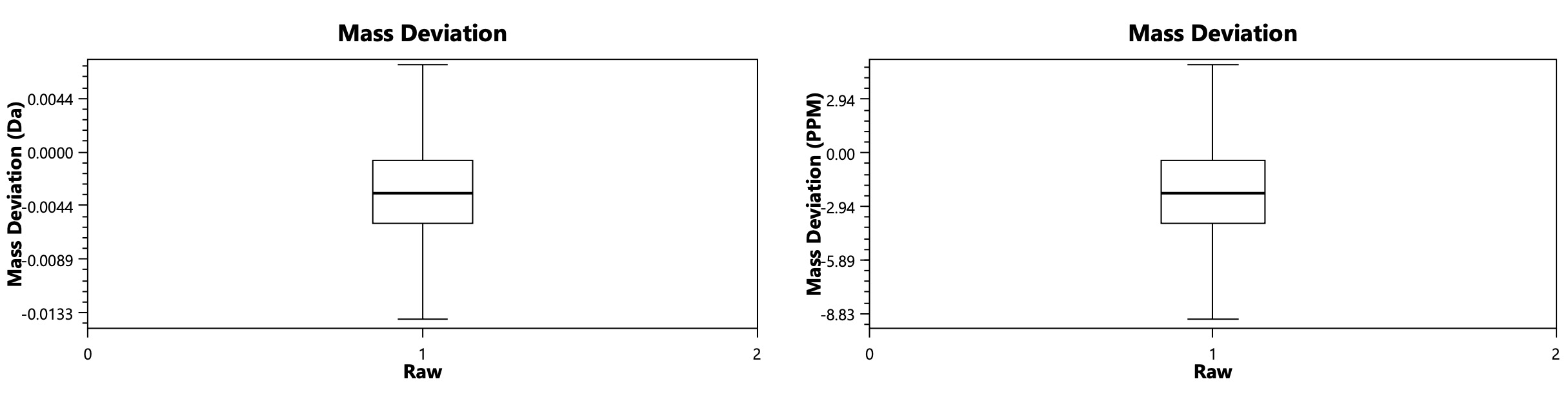
Mass Deviation (PPM)和Mass Deviation (Da)的区别
| 项目 | Mass Deviation (Da) | Mass Deviation (ppm) |
|---|---|---|
| 单位 | Dalton(道尔顿,绝对质量差) | parts-per-million(百万分之一,相对误差) |
| 定义 | 实测质量与理论质量之差:Δm = m实测 − m理论 | 将质量差按理论质量归一化:Δm / m理论 × 10⁶ |
| 数值特点 | 同一数据集中,Da 值随分子量增大而增大 | 同一数据集中,ppm 值基本恒定,与分子量无关 |
| 举例 | 理论 1000.0000 Da 的肽段,实测 1000.0020 Da → Δm = 0.0020 Da | 同上:0.0020 / 1000.0000 × 10⁶ = 2 ppm |
| 适用场景 | 直观查看绝对误差,便于仪器调谐,低分子量时更敏感 | 跨不同质量段比较误差,统一标准,常用于发表 |
| 报告中的位置 | 图表中常标注为 “Mass Deviation (Da)” | 常标注为 “Mass Deviation (ppm)” |
Da 是绝对误差,ppm 是相对误差;ppm 便于不同质量段间横向比较,Da 更直观反映仪器绝对精度。
Score Distribution
Number of PSMs:有多少张图谱被成功匹配到了某个肽段序列上
| 术语 | 中文 | 本质 | 作用 |
|---|---|---|---|
| Target | 真实库 | 包含真实的蛋白质序列(如细菌蛋白 FASTA 文件) | 用于寻找真实肽段 |
| Decoy | 诱饵库 | 由真实序列反向、打乱或随机生成的假蛋白质序列 | 用于估计假阳性 |
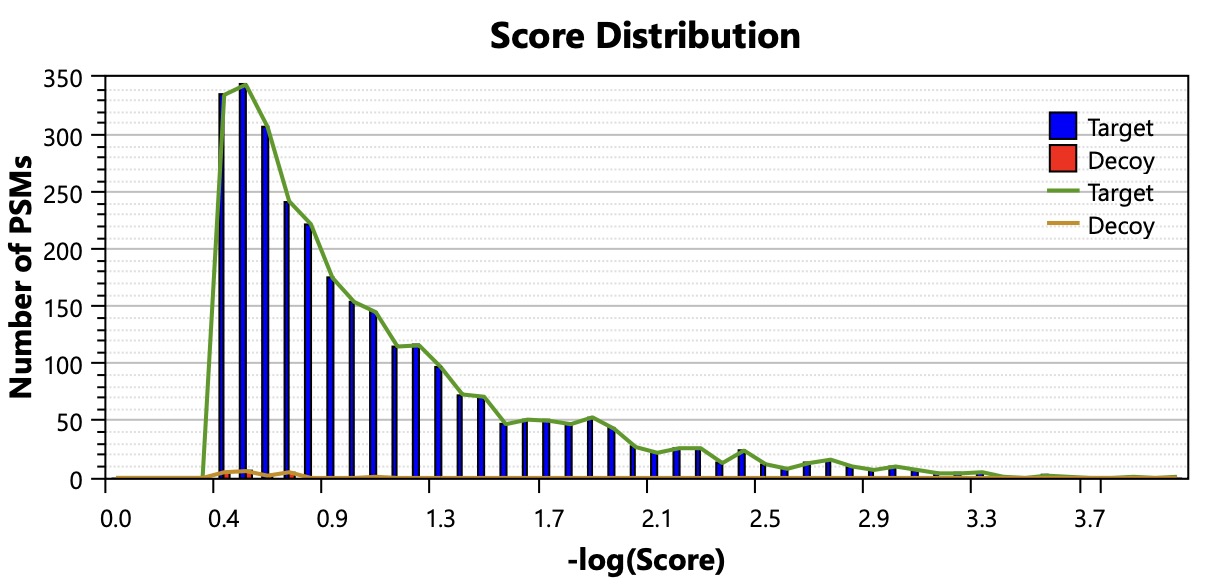
Target FDR 曲线:基于真实库鉴定结果 Decoy FDR 曲线:基于假库鉴定结果
质谱搜库时,总会有一部分图谱错误匹配到数据库中的序列。 这些错误匹配中,有多少是“假阳性”? 通过引入 Decoy(假序列),可以估计: 假阳性率(FDR) = Decoy PSMs / Target PSMs
举例说明: 假设一次搜库后: Target PSMs = 29,460(匹配到真实肽段) Decoy PSMs = 300(匹配到假肽段) 则: FDR = 300 / (29,460 + 300) ≈ 1.0%
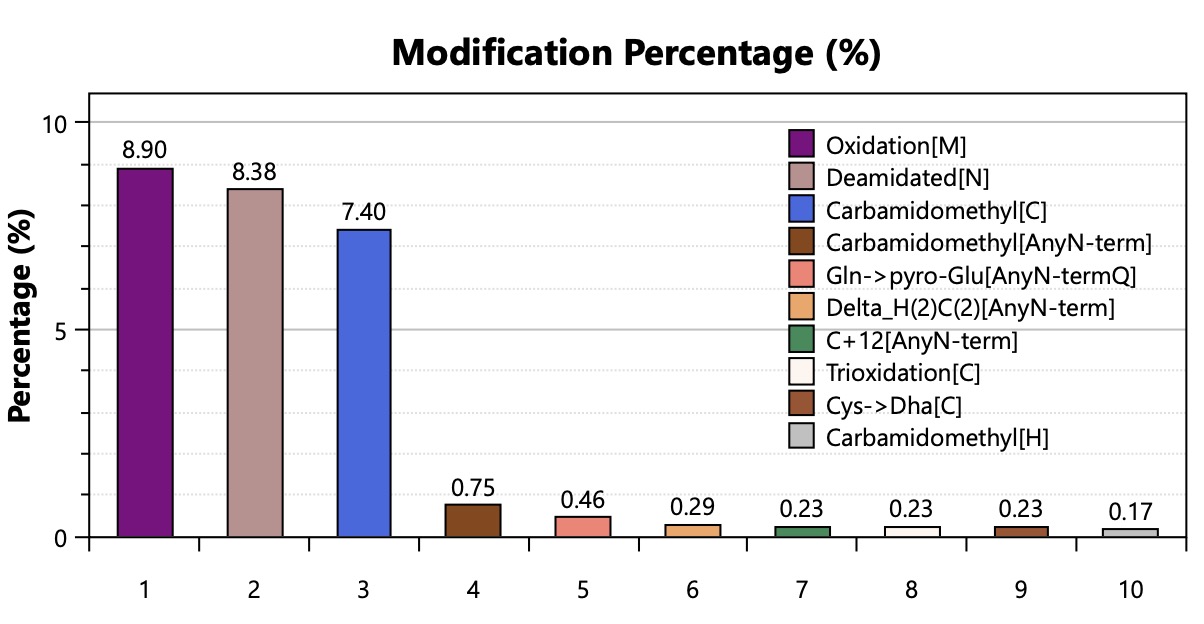
修饰与肽段特征
| 修饰类型 | 比例 | 解读 |
|---|---|---|
| Oxidation[M](甲硫氨酸氧化) | ~8.9% | 较高,提示样本老化或强氧化环境,符合沙漠强UV+干旱条件。 |
| Deamidated[N](天冬酰胺脱酰胺) | ~8.4% | 也是蛋白老化/环境损伤标志。 |
| Carbamidomethyl[C] | ~7.4% | 烷基化修饰,实验处理引入,正常。 |
| 其他稀有修饰 | <1% | 包括焦谷氨酰化、Cys->Dha(脱氢丙氨酸)等,可能是自然降解产物或翻译后修饰。 |
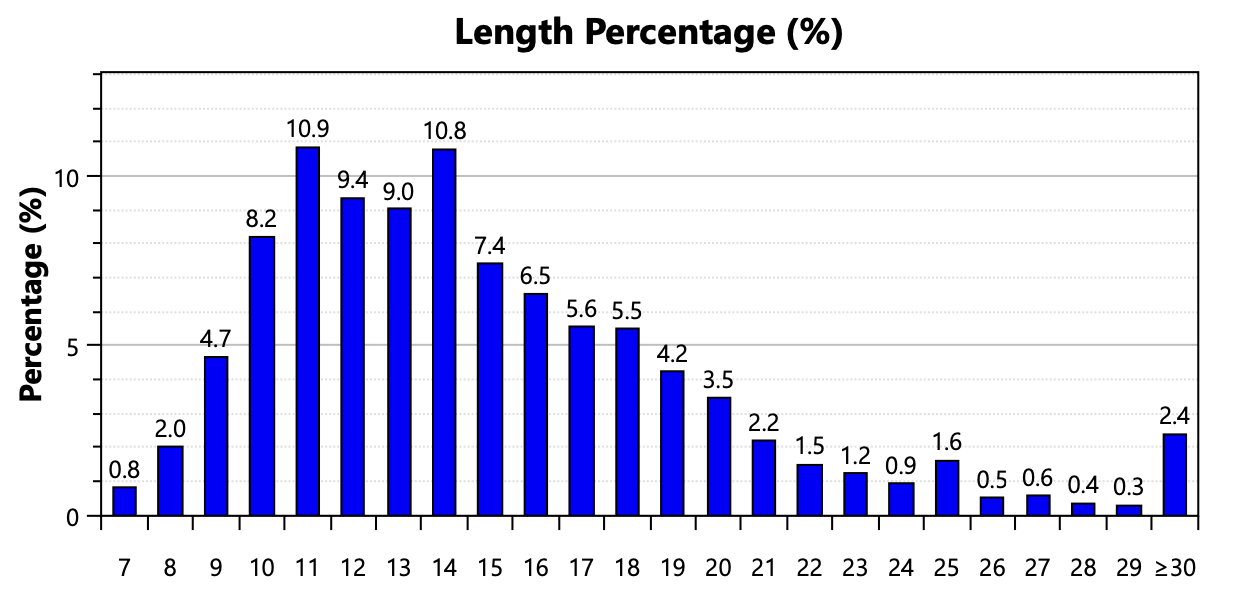
肽段长度与漏切
| 长度区间 | 比例 | 解读 |
|---|---|---|
| 7~30氨基酸 | 占绝大多数 | 符合自然降解肽段特征,非酶解条件下蛋白会随机断裂成中等长度肽段。 |
| >30或<7 | 极少 | 太长可能电离效率低,太短可能无法唯一匹配。 |
ID Rate(鉴定率)
- ID Rate:约 29.37%,在非酶切模式下属于中等偏上水平,说明样本复杂度适中,质谱数据质量良好。
混合谱(Mixed Spectra)
100%混合谱 → 说明很多MS/MS谱图来自共碎片或低纯度母离子,这在复杂环境样本中很常见,尤其是非酶解+低蛋白量条件下。
missed cleavage
100%为0 missed cleavage → 因为是NoEnzyme,没有酶切位点概念,这个指标在这里无意义。
总结
这份报告展示了一次成功的非酶切蛋白质组学分析,数据质量高,修饰控制合理,鉴定结果可信。适用于:
- 未知修饰发现
- 细菌蛋白组全景分析
- 低丰度蛋白/肽段挖掘