数据格式
数据来源:中科新生命,极微量蛋白质组学
从文件夹中的文件扩展名和结构来看,可以初步判断这是Bruker timsTOF 系列质谱仪采集的数据,具体可能是 timsTOF Pro 或 timsTOF fleX 平台。
依据
- tdf 和 .tdf_bin 文件:这是 Bruker timsTOF 系列专属的 TIMS(Trapped Ion Mobility Spectrometry)数据格式,.tdf 是主索引文件,tdf_bin 是原始二进制数据。
- mcf 和 .mcf_idx 文件:这是 Bruker 的 Mass Spectrometry Container Format,用于存储实验配置和元数据。
- mgf 文件:虽然 MGF(Mascot Generic Format)是通用格式,但在 Bruker 系统中常作为导出格式出现。
- analysis.0.DataAnalysis.method 和 .result_c 文件:这是 Bruker Compass 数据分析软件(如 DataAnalysis)生成的文件,进一步支持Bruker 平台。
- sqlite 系列文件:Bruker 的色谱-质谱联用系统常用 SQLite 数据库来存储色谱数据。
这是一套来自 Bruker timsTOF 平台(很可能是 timsTOF Pro)的 LC-MS/MS 数据,结合了离子淌度分离和高分辨质谱,常用于蛋白质组学、代谢组学等领域。
pFind使用
由于MaxQuant在使用过程中总是报有error,改用pFind,一路通畅
pFInd使用视频
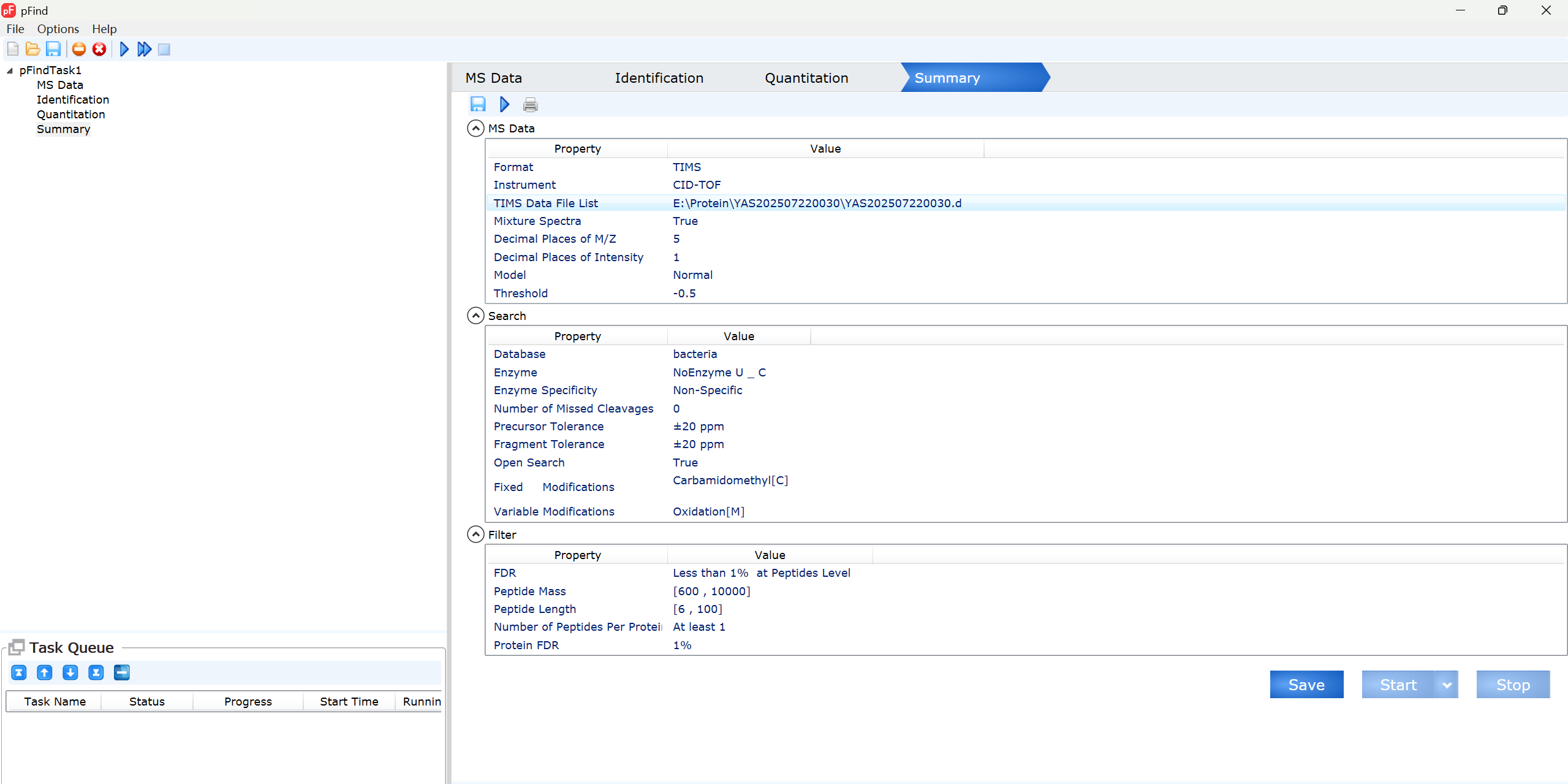
MS Data Format和MS Instrument根据质谱仪填写
timsTOF 默认使用 PASEF(Parallel Accumulation–Serial Fragmentation),但碎裂方式仍是 CID。
Data Extraction(数据提取)
✅ Place of Decimal(小数位)
- M/Z:质荷比的小数位精度设置。
- Intensity:强度值的小数位精度,默认是 1,表示保留一位小数。
✅ Precursor Score(母离子打分模型)
- Model: Normal
- 使用标准模型对母离子进行打分,用于判断 MS1 中提取的母离子是否可信。
- Threshold: -0.5
- 打分阈值,低于这个值的母离子会被过滤掉。负值表示宽松过滤,保留更多候选离子。
✅ Mixture Spectra(混合谱图)
- 含义:是否允许一个 MS2 谱图对应多个母离子(即混合谱)。
- 当前未勾选:说明默认使用“单一母离子”模式,适合干净的数据。
- 建议:如果是复杂样本(如血浆、组织),可以勾选以提高鉴定率。
✅ Output Files(输出文件类型)
- MS1/MS2:输出原始谱图。
- PFB/PFC:pFind 的索引文件,用于后续搜索。
- MGF:导出为通用格式,可用于 Mascot、MaxQuant 等其他软件。
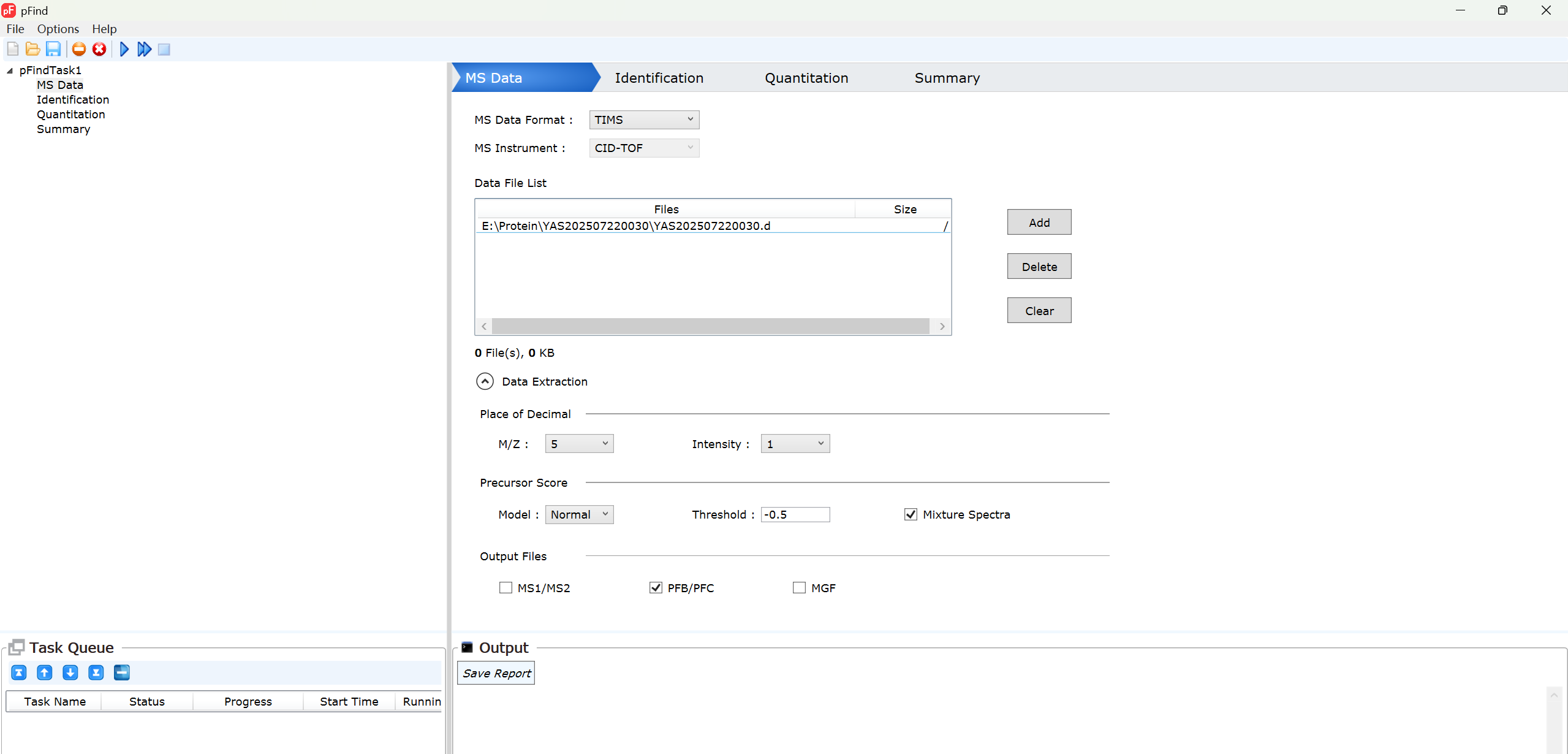
✅ Enzyme(酶切方式)
- 选择:NoEnzyme + Non-Specific
- 含义:不指定酶切位点,允许任意位置断裂。
适用场景: - 样本未经过胰蛋白酶(Trypsin)等酶切处理;
- 或者是 全蛋白质谱(top-down);
- 也可能是 化学裂解 或 非特异性酶切。
风险:搜索空间极大,假阳性风险高,FDR 控制要更严格。
✅ Missed Cleavages(漏切位点数)
- 设置:Up to 0
- 表示 不允许漏切,即每个肽段必须是完整断裂。
- 因为是 NoEnzyme,这个参数无效,因为没有酶切位点。
✅ Mass Tolerance(质量误差)
- 默认即可
- timsTOF 的 MS1 和 MS2 质量精度都在 5–20 ppm 范围内
✅ Fixed Modifications(固定修饰)
- Carbamidomethyl[C] 半胱氨酸烷基化(IAA 处理),防止二硫键形成,标准固定修饰。
✅ Variable Modifications(可变修饰)
| 修饰 | 含义 |
|---|---|
| Acetyl[Any N-term] | 蛋白 N 端乙酰化 |
| Acetyl[K] | 赖氨酸乙酰化 |
| Oxidation[M] | 甲硫氨酸氧化 |
| Amidated [Any C-term] | C 端酰胺化 |
| Carbamyl [K/Any N-term] | 尿素诱导的氨基甲酰化 |
| Cation:Na [Any C-term] | 钠离子加合物 |
Oxidation[M]为参考视频中推荐
✅ Result Filter(结果过滤)
- 默认即可
| 参数 | 值 | 含义 |
|---|---|---|
| FDR ≤ 1% | 假发现率控制在 1% 以内,标准过滤阈值 | |
| Peptide Mass Range | 600–10000 Da | 过滤掉过小或过大的肽段,避免异常值 |
| Min Peptides per Protein | ≥1 | 每个蛋白至少有一条肽段支持 |
| Show Proteins / Peptides | 显示蛋白或肽段级别的结果 |
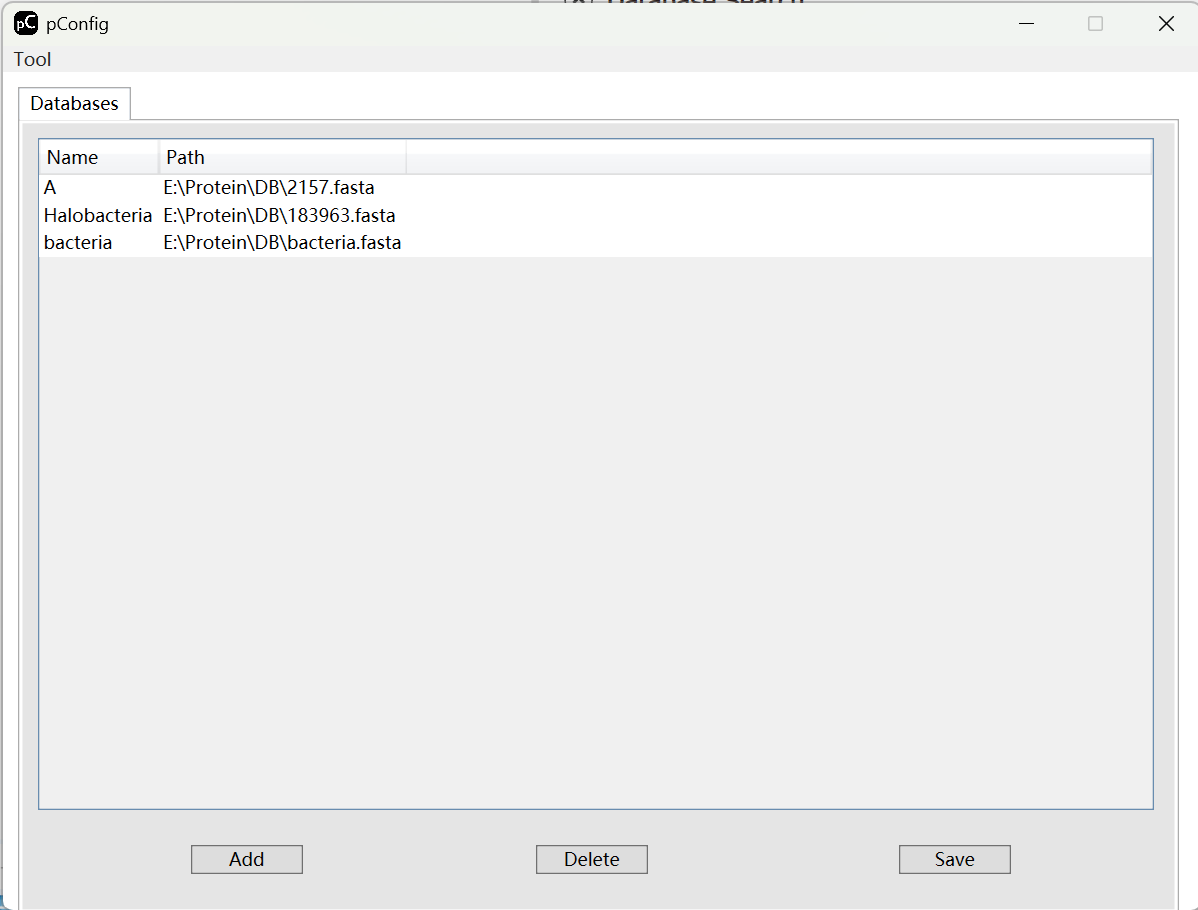
数据库的选择与导入,待搜库物种蛋白质序列在Uniprot中下载。
主要在Taxonomy中查询后,download人工审核过的数据也就是Reviewed (Swiss-Prot)即可。
待搜库物种蛋白质序列下载方法
如果勾选Add contaminant会把各种污染库也包括进去,在我的数据中,add后测出了大量污染蛋白……
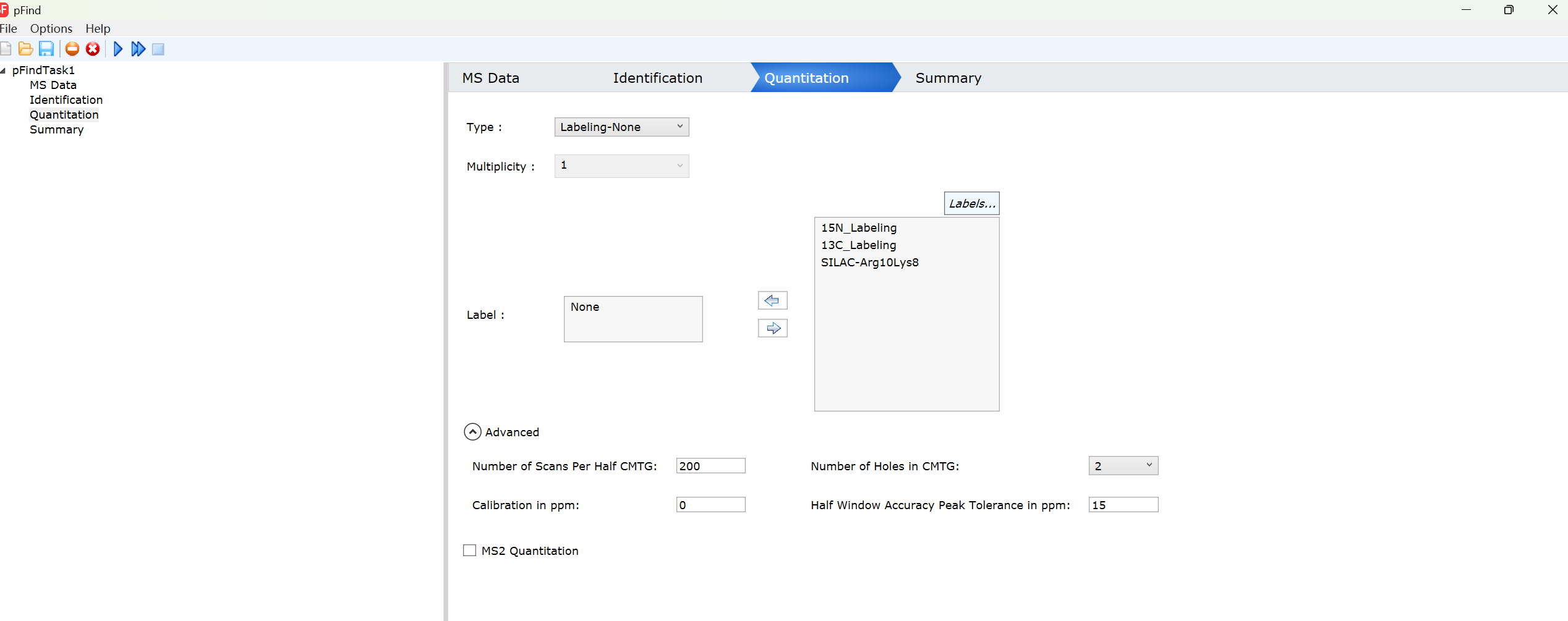
✅ Type(定量类型)
- 当前选择:Labeling-None
- 含义:不使用任何同位素标记进行定量(即 非标记定量)。
- 适用场景:DIA(如 diaPASEF)、label-free、DDA 非标记定量。
✅ Multiplicity(标记类型选择) 下面仍然列出了可选的标记类型,这些是备用选项,不影响当前结果:
| 选项 | 含义 |
|---|---|
| 15N_Labeling | 全氮15标记(如细菌培养在15N培养基中) |
| 13C_Labeling | 全碳13标记 |
| SILAC-Arg10Lys8 | 经典 SILAC 标记,重标精氨酸(+10 Da)和赖氨酸(+8 Da) |
默认即可
✅ Number of Scans Per Half CMTG
✅ Number of Holes in CMTG
✅ Calibration in ppm
- timsTOF 本身精度高,通常不需要手动校正。
✅ Half Window Accuracy Peak Tolerance in ppm
- timsTOF 的 MS1 精度在 5–20 ppm,15 ppm 是保守且安全的设置。
Save后Start
Kimi分析结果文件
细菌&古菌
噬盐菌(Halophiles)代表性蛋白发现
| 蛋白名称 | 物种来源 | 功能/特征 | 备注 |
|---|---|---|---|
| GLPK_HALSA | Halobacterium salinarum | 甘油激酶(glycerol kinase) | 经典嗜盐古菌,适应高盐环境 |
| GLPK_HALMA | Haloarcula marismortui | 甘油激酶 | 极端嗜盐菌,分离自死海 |
| SODF_METTH | Methanothermobacter thermautotrophicus | 超氧化物歧化酶 | 嗜盐兼嗜热,常见于盐湖沉积物 |
噬热菌(Thermophiles)代表性蛋白发现
| 蛋白名称 | 物种来源 | 功能/特征 | 备注 |
|---|---|---|---|
| EFTU_METCA | Methylococcus capsulatus | 延伸因子 Tu | 嗜热甲烷氧化菌,常见于热泉或地热区 |
| EFTU_THIDL | Thiomonas delicata | 延伸因子 Tu | 嗜热硫氧化菌,适应高温酸性环境 |
| EFTU_METPP | Methylibium petroleiphilum | 延伸因子 Tu | 嗜热兼耐石油污染菌,常见于热液系统 |
这些蛋白的发现支持了阿塔卡马沙漠土壤中存在极端环境微生物的假设,并可为后续研究其干旱适应机制、盐胁迫响应或热稳定性蛋白功能提供线索。
Proteobacteria 在整个细菌库中占比 >60%,是阿塔卡马沙漠样本中最活跃的细菌门类。
Actinobacteria虽然数量不如 Proteobacteria 多,但也被明确检测到,且多为耐旱、耐辐射的代表菌属。
Fungi
真菌种类明确,均为极端环境适应型
| 真菌种类 | 生态意义 |
|---|---|
| Neurospora crassa | 模式真菌,耐干旱、紫外线 |
| Schizosaccharomyces pombe | 耐高盐、氧化应激 |
| Candida albicans | 机会致病,可能来自动物或人类污染 |
| Komagataella phaffii (Pichia pastoris) | 工业酵母,耐极端pH、渗透压 |
| Debaryomyces hansenii | 耐高盐真菌,常见于盐湖、沙漠土壤 |
| Aspergillus fumigatus / oryzae | 耐干旱、产孢子能力强 |
这些真菌均为极端环境适应型,与阿塔卡马沙漠的高盐、干旱、强紫外线条件高度匹配。
功能模块高度集中于“能量代谢 + 抗氧化 + 蛋白稳态”
| 功能通路 | 代表蛋白 | 生态适应意义 |
|---|---|---|
| 线粒体能量代谢 | ATPB(ATP合成酶β亚基)、EF-Tu、EF-G | 在极端干旱中维持能量供应 |
| 抗氧化应激 | SOD(超氧化物歧化酶) | 对抗强紫外线诱导的ROS |
| 蛋白质稳态 | Ubiquitin、Ribosomal fusion proteins | 维持蛋白折叠与降解平衡 |
| 甲醇代谢 | AOX1/2(酒精氧化酶) | 可能利用沙漠中微量甲醇或甲烷 |
生态学意义(结合阿塔卡马沙漠)
| 沙漠特征 | 真菌蛋白响应 |
|---|---|
| 极端干旱 | 高表达ATP合成酶、EF-Tu → 维持能量效率 |
| 高盐环境 | Debaryomyces hansenii(耐盐)→ 利用Na⁺/K⁺泵 |
| 强紫外线 | SOD(抗氧化)→ 清除ROS,保护DNA/蛋白 |
| 低营养 | AOX1/2 → 可代谢痕量甲醇、甲烷或挥发性有机物 |
该真菌蛋白组清晰地反映了阿塔卡马沙漠极端环境下真菌的能量代谢与抗氧化适应机制,无人类污染,是研究极端生态微生物适应的优质样本。